研究内容
- ヒトがん細胞パネル(JFCR39)を用いた薬剤感受性―マルチオミックス統合データベース(JFCR39-DB)の開発と抗がん剤スクリーニング
- 新規PI3K阻害剤ZSTK474の同定と創薬研究
- ゴルジ体を標的とした新規抗がん物質の同定と抗がん分子機序の解析
1. ヒトがん細胞パネル(JFCR39)を用いた薬剤感受性―マルチオミックス統合データベース(JFCR39-DB)の開発と抗がん剤スクリーニング
がんが日本人の死因のトップを占めるようになって久しい。がんの薬物療法は、外科的手術、放射線療法と並ぶ重要な治療法である。転移を伴う進行がんや血液腫瘍など、他の治療法を用いることができないことも多く、がんの薬物療法に対する期待は大きい。1990年代までの抗がん剤の多くは、細胞増殖に伴うDNA合成、細胞分裂を標的とする化学療法剤であり、血液腫瘍や一部の固形癌を除き、満足な治療成績は残せなかった。ところが、20世紀後半の分子生物学や遺伝医学的研究手法の進歩により、がんの分子レベルでの理解が進んだ結果、がんで特異的に活性化している分子を標的とした抗がん剤(分子標的薬)が2000年代以降に次々と開発され、高い治療効果かつ低毒性を実現している。一方、分子標的薬に耐性を獲得したがんや、そもそも分子標的薬の開発が進んでいないがんなど、有効な治療法が存在しないがんは多数残されている。このようなアンメットメディカルニーズに応える新たな治療標的分子の同定と、その阻害剤の開発が求められている。
新たながんの治療標的分子を見出すアプローチとして通常用いられるのは、前述した分子生物学や遺伝医学的なアプローチによりがんの特性を解析して同定された、がんで特異的に変異ないし活性化している分子の中から、薬剤の標的となりうる(ドラッガブルな)分子を選抜する方法である。HanahanとWeinbergは、共著のレビュー”Hallmarks of Cancer: The Next Generation”において、増殖シグナルの活性化、細胞周期制御の異常、血管新生、転移浸潤や、最近注目を浴びる免疫逃避機構など、10種類のがんの特徴を挙げている(1)。これらの標的の一部は実際に阻害剤が開発・薬事承認され、高い治療効果を発揮している。一方、我々は、新たながんの治療標的分子を見出す別の方法として、化合物を起点としたアプローチをとっている。この方法をとるにあたり、注目すべきユニークな抗がんスペクトルを持つ化合物をいかに効率よく同定するか、が重要となる。我々は、JFCR39と呼ばれる39種のヒトがん細胞株からなるパネルを利用したがん細胞パネル法を導入している。
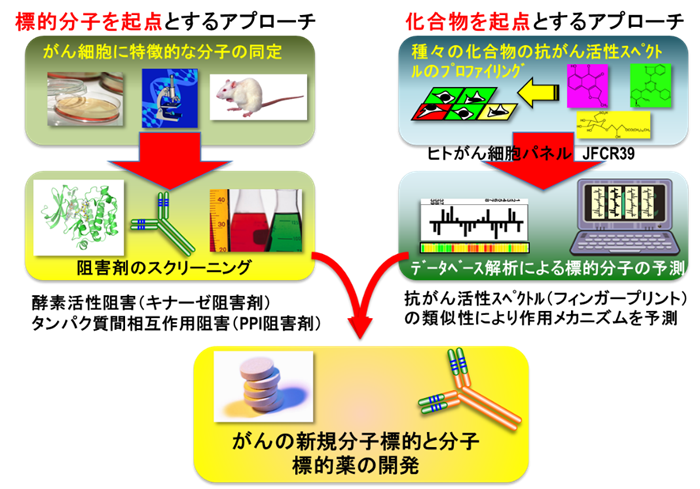
がん細胞パネル法は、1990年前後にアメリカ国立がん研究所(NCI)にて60種のヒトがん細胞(NCI60)を用いて開発された抗がん剤スクリーニング法である。JFCR39パネルは、NCIのShoemaker博士らの全面協力の下、当部の初代部長である矢守隆夫博士が1990年代にがん研究会(JFCR)にて独自に立ち上げた(2,3)。本手法は、既知の抗がん剤や阻害剤について、JFCR39に含まれる各細胞株に対する抗がん活性(有効濃度)を測定し、細胞株毎に異なる有効濃度のパターン(フィンガープリントと呼ぶ)としてデータベース(DB)化することにより、新規物質の抗がんメカニズムをフィンガープリントの類似性により予測しようとするものである。当部ではこれまでに、数千種類の化合物についてJFCR39に対する抗がん活性データを測定しており、これまでに日米で薬事承認された化学療法剤や分子標的薬の大半を網羅しているばかりでなく、現在世界中の製薬会社で臨床試験、ないし、非臨床試験で評価中の抗がん物質についても、入手可能なものから順に測定してDBに加えてきた。このJFCR39に対する化合物の抗がん活性データベースをレファレンスに用いることにより、作用メカニズムが未知の抗がん物質に関する抗がんメカニズムの推定をすることが可能である。もう少し具体的に言うと、未知物質についてJFCR39に対する抗がん活性フィンガープリントを測定し、それを取り貯めたレファレンス化合物のフィンガープリントと比較することにより、既存物質のどれと作用メカニズムが類似しているか、または、既存物質のどれとも異なるユニークな抗がんメカニズムを示すものなのかを評価することが可能である。本研究基盤を利用して、日本国内のアカデミア研究者への支援研究、製薬企業との共同研究を展開しており、新規PI3K阻害剤であるZSTK474や、新規ゴルジ体機能阻害剤AMF-26/M-COPAを同定することに成功した(4,5)。
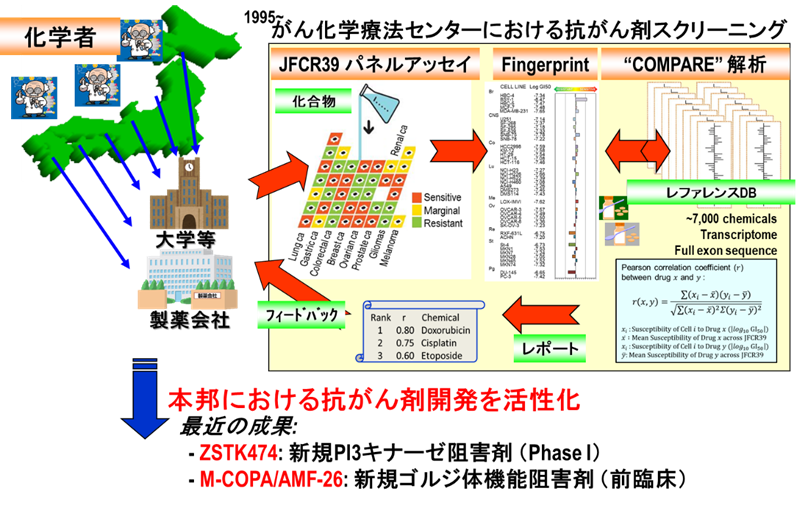
さらに興味深いことに、ある特定の作用メカニズムの抗がん物質として同定された化合物が、そのフィンガープリントから、メインターゲットとは異なる標的分子(オフターゲット)が同定されたり、場合によっては、むしろそれがメインターゲットであることが判明した例もある(6)。以上のように、標的特異性が高い化合物もあれば、複数の標的を阻害する化合物もあり、レファレンス化合物とのフィンガープリント比較により、細かな特性の違いとして現れる。このような細胞株毎に異なる抗がん活性は、パネルに含まれるがん細胞株の遺伝的背景の違いを反映していると考えられる。このことから、我々は、JFCR39に関してゲノムワイドな遺伝子発現、変異、タンパク質発現などさまざまなデータを取得し、マルチオミックスデータとしてデータベース化し、薬剤感受性データと統合データベース化(JFCR39-DB)して相互比較することが可能となっている(7,8)。このJFCR39-DBを利用して、薬剤感受性と関連する遺伝子発現や変異、細胞内シグナルパスウェイ活性化状態などを探索し、薬剤感受性予測バイオマーカーの同定や、薬剤感受性の原因となる分子背景の解明に役立てている。
当研究室では、文部科学省・新学術領域研究がサポートする支援活動として、がん細胞パネル用いた化合物の分子プロファイリング支援を担当している。ご興味のある方は、下記ホームページを参照されたい。
文部科学省 新学術領域研究 先端モデル動物支援プラットフォーム・分子プロファイリング支援活動
参考文献
- Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell. 2011 Mar 4;144(5):646-74. doi: 10.1016/j.cell.2011.02.013. Review.
- Shoemaker RH. The NCI60 human tumour cell line anticancer drug screen. Nat Rev Cancer. 2006 Oct;6(10):813-23. Review.
- Yamori T, Matsunaga A, Sato S, Yamazaki K, Komi A, Ishizu K, Mita I, Edatsugi H, Matsuba Y, Takezawa K, Nakanishi O, Kohno H, Nakajima Y, Komatsu H, Andoh T, Tsuruo T. Potent antitumor activity of MS-247, a novel DNA minor groove binder, evaluated by an in vitro and in vivo human cancer cell line panel. Cancer Res. 1999 Aug 15;59(16):4042-9.
- Yaguchi S, Fukui Y, Koshimizu I, Yoshimi H, Matsuno T, Gouda H, Hirono S, Yamazaki K, Yamori T. Antitumor activity of ZSTK474, a new phosphatidylinositol 3-kinase inhibitor. J Natl Cancer Inst. 2006 Apr 19;98(8):545-56.
- Ohashi Y, Iijima H, Yamaotsu N, Yamazaki K, Sato S, Okamura M, Sugimoto K, Dan S, Hirono S, Yamori T. AMF-26, a novel inhibitor of the Golgi system, targeting ADP-ribosylation factor 1 (Arf1) with potential for cancer therapy. J Biol Chem. 2012 Feb 3;287(6):3885-97.
- Katayama R, Aoyama A, Yamori T, Qi J, Oh-hara T, Song Y, Engelman JA, Fujita N. Cytotoxic activity of tivantinib (ARQ 197) is not due solely to c-MET inhibition. Cancer Res. 2013 May 15;73(10):3087-96.
- Dan S, Tsunoda T, Kitahara O, Yanagawa R, Zembutsu H, Katagiri T, Yamazaki K, Nakamura Y, Yamori T. An integrated database of chemosensitivity to 55 anticancer drugs and gene expression profiles of 39 human cancer cell lines. Cancer Res. 2002 Feb 15;62(4):1139-47.
- Dan S, Okamura M, Seki M, Yamazaki K, Sugita H, Okui M, Mukai Y, Nishimura H, Asaka R, Nomura K, Ishikawa Y, Yamori T. Correlating phosphatidylinositol 3-kinase inhibitor efficacy with signaling pathway status: in silico and biological evaluations. Cancer Res. 2010 Jun 15;70(12):4982-94.